Rechercher
Un modèle inédit du mécanisme de ségrégation active des chromosomes bactériens
publié le

L’équipe de Jean-Yves Bouet au Laboratoire de microbiologie et génétique moléculaires, en collaboration avec des équipes du Centre de biochimie structurale et du Laboratoire Charles Coulomb, révèle le mécanisme d’assemblage dynamique et de confinement de l’appareil de ségrégation des chromosomes chez les bactéries. Ces travaux sont publiés dans la revue Cell Systems.
Le génome des bactéries est composé d’un ou plusieurs chromosomes et d’une quantité variable de petites molécules d’ADN autonomes appelées « plasmides ». Ces derniers permettent aux bactéries de survivre et de se répandre dans des niches écologiques particulières, et sont très souvent porteurs des gènes de virulence ou de résistance aux antibiotiques. Après leur duplication, l’ensemble des différentes molécules d’ADN sont fidèlement transmises à chacune des deux cellules filles lors de l’étape de ségrégation.
Chez les bactéries, la ségrégation des chromosomes et des plasmides est assurée par un système actif très efficace mais minimaliste qui se compose de seulement trois éléments essentiels : un centromère, une protéine de fixation au centromère (ParB) et une protéine qui hydrolyse l’ATP (ParA), considérée comme la protéine motrice de la ségrégation. L’architecture du complexe nucléoprotéique, appelé complexe de partition, qui se forme sur le centromère, reste mal comprise et le mécanisme moléculaire conduisant à cet assemblage était controversé avec deux modèles principaux en concurrence. Un premier modèle proposait que les protéines ParB polymérisent uni-dimensionnellement sur de longues distances le long de l’ADN à partir du centromère. Un second modèle postulait que cette polymérisation est limitée et initiée à la fois à partir du centromère et d’ADN non-centromériques permettant des pontages entre ces zones de polymérisation limitée. Ces deux modèles suggéraient un assemblage organisé et structuré du complexe de partition avec lequel interagit l’ATPase ParA.
Les chercheurs de l’équipe de Jean-Yves Bouet au Laboratoire de Microbiologie et Génétique Moléculaires, en collaboration avec les équipes de Marcelo Nollmann au Centre de Biochimie Structurale (Montpellier) et d’Andrea Parmeggiani au Laboratoire Charles Coulomb (Montpellier) ont étudié le mécanisme de formation du complexe de partition du plasmide F d’Escherichia coli en combinant des approches de séquençage à haut-débit à l’échelle du génome entier, de biochimie, de microscopie à super-résolution et de modélisation physique. Les résultats permettent de montrer que l’ensemble des protéines ParB (environ 1000 molécules par cellule) sont activement confinées autour du centromère en se fixant de façon stochastique à l’ADN spatialement proche, grâce à plusieurs interactions synergiques, protéine-protéine et protéine-ADN.
Ces travaux permettent de proposer un nouveau modèle, dit de « nucléation et confinement », basé sur l’auto-assemblage de la protéine ParB qui forme un réseau dynamique sur l’ADN autour du centromère. Il révèle que l’architecture du complexe de partition n’est pas ordonnée mais fortement dynamique. Ces travaux ouvrent des perspectives nouvelles pour comprendre au niveau moléculaire le mécanisme actif de ségrégation de l’ADN chez les bactéries. De plus, ce modèle d’assemblage et les approches utilisées pour caractériser à grande échelle les interactions dynamique entre macromolécules peuvent être généralisables à un grand nombre de machineries moléculaires qui s’auto-assemblent en superstructures fonctionnelles.
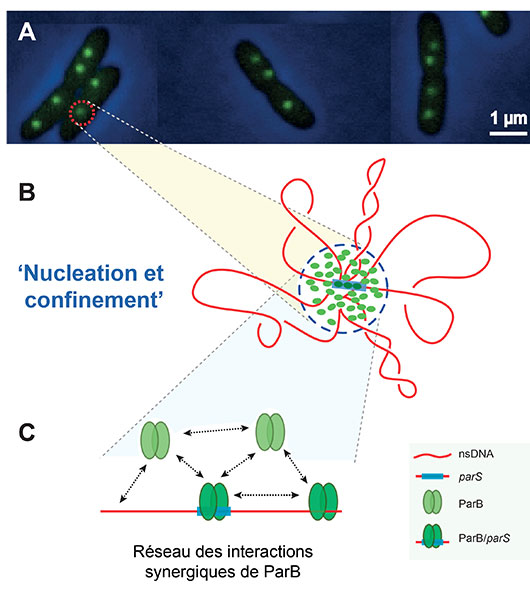
© Jean-Yves Bouet
Figure : Modèle moléculaire d’assemblage du complexe de partition chez les bactéries. (A) Exemples de cellules d’Escherichia coli visualisées par microscopie à fluorescence. Les spots verts correspondent à la fluorescence émise par les molécules ParB-mVenus et permettent de visualiser le complexe de partition. (B) Zoom représentant schématiquement le modèle de « Nucléation et confinement » dans lequel la fixation spécifique de molécules de ParB induit la nucléation de l’ensemble des autres molécules de ParB présentes dans la cellule en les confinant de façon active autour du centromère par interaction stochastique avec l’ADN avoisinant. (C) Représentation schématique de l’ensemble des interactions synergiques de ParB nécessaires au confinement de l’ensemble des ParB autour du centromère.
© Jean-Yves Bouet
Publication :
Stochastic self-assembly of ParB proteins builds the bacterial DNA segregation apparatus, Aurore Sanchez, Diego I. Cattoni, Jean-Charles Walter, Jérôme Rech, Andrea Parmeggiani, Marcelo Nollmann and Jean-Yves Bouet
Cell Systems (2015). Vol. 1, Issue 2, p163–173
doi : 10.1016/j.cels.2015.07.013
HAL : hal-01191677
Contact chercheur
Jean-Yves Bouet
Laboratoire de microbiologie et génétique moléculaires (LMGM)
CNRS UMR5100, Université Toulouse III – Paul Sabatier
118 Route de Narbonne
31062 Toulouse Cedex 9
Tél : 05 61 33 59 06








